机器学习之分类(含决策树,SVM,knn,bayes)
今天晚上趁着云计算选修课的时间来更新一波。。。。。。
最近一直没啥时间,在做自然语言处理,HDR相关的一些工作(之后有机会会更新),这次试验有点小小的仓促。唉,不得不说,大三,属实开始忙碌起来了。
这次的内容包含四个基本的分类算法。其中,决策树,knn为调用库函数方法,SVM,bayes为手写代码。本博文重在代码实现,笔者对其思想不做过多描述,建议最好在了解其方法思想进行代码实现时观看。
笔者依旧来给实验报告打个草稿,也给各位看官一些参考。
笔者在整理资料时,在平台上参考了不少资料,在此表示感谢。
笔者代码已经经过验证,思路清晰,结果可靠。
笔者强调一下,解决问题的思想是很重要的,写代码前一定要吃透其思想,这样无论多么复杂的程序最终都能够写出来,其花费的时间也不过是调py语法错误的时间而已。
一.实验内容:
实验目的
1. 掌握主要分类算法的基本原理与实现。
2. 比较不同分类算法的结果,分析其优缺点
实验问题背景
根据美国疾病控制预防中心统计,现在美国1/7的成年人患有糖尿病。到2050年,这个比例将会增长至1/3。据分析,是否糖尿病患者与怀孕次数,血糖、血压、皮脂厚度、胰岛素、BMI身体质量指数、糖尿病遗传函数、年龄等特征密切相关。通过机器学习预测是否患有糖尿病,具有非常大的应用价值。
实验问题描述
现有一份糖尿病患者数据集diabetes.csv,该数据集有768个数据样本,每个样本有8个特征和一个类别标签,具体信息如下:
| 特征名称 | 特征含义 | 取值举例 |
| Pregnancies | 怀孕次数 | 6 |
| Glucose | 2小时口服葡萄糖耐受实验中的血浆葡萄浓度 | 148 |
| BloodPressure | 舒张压 (mm Hg) | 72 |
| SkinThickness | 三头肌皮褶厚度(mm) | 35 |
| Insulin | 2小时血清胰岛素浓度 (mu U/ml) | 0 |
| BMI | 体重指数(weight in kg/(height in m)^2) | 33.6 |
| DiabetesPedigree Function | 糖尿病谱系功能(Diabetes pedigree function) | 0.627 |
| Age | 年龄 | 50 |
| class | 是否患有糖尿病 | 1:阳性;0:阴性 |
实验要求
- 对数据做适当必要的预处理,将数据集按照3:1分为训练集和验证集。
- 运用决策树、支持向量机、近邻法、贝叶斯等分类算法(至少实现其中两种)实现分类。
- 比较不同分类算法的结果,分析其优缺点。
- 分析算法实现中关键参数的影响,以曲线等可视化的方式展示。
实验报告内容
1. 实验内容:
2. 主要算法的程序实现:
3. 调试报告:调试过程中遇到的主要问题是如何解决的。
4.实验结果比较分析。
5. 实验总结:对实验用到的理论知识的理解,在方法设计上有何创新。
实验报告提交要求
1. 实验报告应使用指定的实验报告的封面,实验名称为实验指导书中的实验名称。
2. 实验报告提交内容包括:1)实验报告的word版;2)程序源代码(单独放在一个文件夹内)。将上述2部分内容放在一个文件夹内。命名格式:班级-学号-姓名
3.实验完成后一周内,在课堂派上提交(直接上传原始文件,不要压缩)。
二:数据预处理:
代码:
"""
coding:utf-8
@author: Li Sentan
@time:2021.10.26
@file:shiyansecondpre.py
"""#数据预处理import pandas as pd
import numpy as np
def readcsv(path):datas = pd.read_csv(path)datas_Glucose = datas.Glucosedatas_length = len(datas_Glucose)head = datas.columnsreturn datas ,datas_Glucose, head ,datas_length
if __name__ == '__main__':path = 'diabetes.csv'datas,datas_Glucose, head ,datas_length = readcsv(path)df = pd.DataFrame(data=datas)Gl = df['Glucose'].valuesGl0 = []Bl = df['BloodPressure'].valuesBl0 = []BMI = df['BMI'].valuesBMI0 = []for i in range(datas_length):if Gl[i] <= 0:Gl0.append(i)Gl[i] = 0if Bl[i] <= 0:Bl0.append(i)Bl[i] = 0if BMI[i] <= 0:BMI0.append(i)BMI[i] = 0Gmean = np.sum(Gl)/(len(Gl)-len(Gl0))Bmean = np.sum(Bl)/(len(Bl)-len(Bl0))BMImean = np.sum(BMI)/(len(Gl)-len(Gl0))for i in Gl0:df.loc[i,'Glucose']= Gmeanfor i in Bl0:df.loc[i,'BloodPressure'] = Bmeanfor i in BMI0:df.loc[i,'BMI'] = BMImeana = np.std(df["Glucose"])b = np.std(df["BloodPressure"])c = np.std(df["BMI"])print("normal area of Glucose:({}--->{})".format(Gmean - 3 * a,Gmean + 3 * a))print("normal area of BloodPressure:({}--->{})".format(Bmean - 3 * b,Bmean + 3 * b))print("normal area of BMI:({}--->{})".format(BMImean - 3 * c,BMImean + 3 * c))for j in df["Glucose"]:if j < Gmean - 3*a or j > Gmean + 3*a:print(j)print("xxxxxxxx")for j in df["BloodPressure"]:if j < Bmean - 3*b or j > Bmean + 3*b:print(j)print("xxxxxxxx")for j in df["BMI"]:if j < BMImean - 3 * c or j > BMImean + 3 * c:print(j)df.to_csv('diabetes2.csv')
原数据文件在笔者资源区可免费下载。
观察数据可知,'Glucose','BloodPressure',"BMI"这三个变量不可能为零,其他的数据咱也没有十分充足的证据说人家不对,毕竟大千世界,无奇不有,有些数据虽是离群点,但还是很真实的。
所以读取数据之后,把相应的不正确的值给它改成相应列的平均值。
然后我还打印出来了3σ范围之内的正常值和3σ之外的特殊值,结果如下:
三:决策树
代码:
"""
coding:utf-8
sorting algorithms:DecisionTreeClassifier
@author: Li Sentan
@time:2021.10.27
@file:shiyansecond1.py
"""from sklearn.preprocessing import StandardScaler
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split
import graphviz
import pydotplus
from sklearn import tree
from IPython.display import Image
import pandas as pd
import numpy as np
import osdata = pd.read_csv("diabetes2.csv")
y = data['Class']
x = data.drop('Class', axis=1)
X = x.drop('Unnamed: 0',axis=1)X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=100, train_size=0.75)
columns = X_train.columns
# ss_X = StandardScaler()
# ss_y = StandardScaler()
# X_train = ss_X.fit_transform(X_train)
# X_test = ss_X.transform(X_test)
model_tree = DecisionTreeClassifier(criterion="gini")
model_tree.fit(X_train, y_train)
y_prob = model_tree.predict_proba(X_test)[:,1]
y_pred = np.where(y_prob > 0.5, 1, 0)
count1 = 0
count2 = 0
j = 0
for i in y_test:if y_pred[j]==i:count1 = count1 +1else:count2 = count2 + 1j = j + 1
print("the accuracy:",count1/len(y_pred))
print(y_pred)
print(y_test)
# print(model_tree.score(X_test, y_pred))
# 可视化树图
data_ = pd.read_csv("diabetes2.csv")
data_feature_name = data_.columns[1:-1]
data_target_name = np.unique(list(map(str,data_["Class"])))os.environ["PATH"] += os.pathsep + 'G:/Graphviz/bin/'
dot_tree = tree.export_graphviz(model_tree,out_file=None,feature_names=data_feature_name,class_names=data_target_name,filled=True, rounded=True,special_characters=True)
graph = pydotplus.graph_from_dot_data(dot_tree)
img = Image(graph.create_png())
graph.write_png("out2.png")笔者在这里调用了sklearn.tree中的DecisionTreeClassifier,首先把数据按照3:1分成训练集和测试集,因为我数据预处理用pandas.Dataframe()生成的diabeters2.csv文件中,增加了index列,
x = data.drop('Class', axis=1)
X = x.drop('Unnamed: 0',axis=1)
所以我通过删除'Class','Unnamed: 0',得到所有属性的列向量X。
model_tree = DecisionTreeClassifier(criterion="gini") #定义model_tree为DecisionTreeClassifier()的一个具体对象,criterion="gini"代表用gini系数#表示分类的不纯性,并以此来作为划分决策树的依据。 model_tree.fit(X_train, y_train) #fit()表示训练拟合模型 y_prob = model_tree.predict_proba(X_test)[:,1] #把X_test数据代入,每个数据预测分类会以概率从小到大得到类型,本数据只有两个类,所以[:,1] #表示选择第二个值,要是写成[:,0],得到模型的准确率正好是原模型的错误率。 y_pred = np.where(y_prob > 0.5, 1, 0) #其实这一步可加可不加,因为上一步已经得到0或1的值了。
接下来就可以得到准确率了。
最后,有一个决策树可视化的方法非常不错:
data_target_name = np.unique(list(map(str,data_["Class"])))
其中map()函数表示将类中的数据都变成str类型,np.unique()方法表示去掉list中重复的字符串。
class——names比较好玩,下面是它的解释:
class_names : list of strings, bool or None, optional (default=None) Names of each of the target classes in ascending numerical order. Only relevant for classification and not supported for multi-output. If True, shows a symbolic representation of the class name.
按升序排列,所以也可写成:
data_target_name = np.unique(['yes','no'])
其中的'yes'和'no'顺序可变,不影响输出结果,因为n在y之前。
最后结果如下图:

最后,提出一个探究性小问题, 训练集测试集比例改变导致模型改变,从而导致准确率改变可以理解,但是,保持训练集测试集比例不变的话,每次运行的结果准确率还是会改变的,这一点确实比较疑惑。笔者猜想,原因可能是每次运行时,系统库中的fit()方法构建函数模型的方式会改变。笔者在之后会进一步探究。
四:SVM
"""
coding:utf-8
sorting algorithms:SVM
@author: Li Sentan
@time:2021.10.27
@file:shiyansecond2.py
"""import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as pltdef readcsv(path):datas = pd.read_csv(path)datas_Pregnancies = datas.Pregnanciesdatas_length = len(datas_Pregnancies)head = datas.columnsreturn datas ,datas_Pregnancies, head ,datas_length
#加载数据
def create_data():path = "diabetes2.csv"datas, datas_Pregnancies, head, datas_length = readcsv(path)# 选取前两类作为数据df = pd.DataFrame(data=datas)X = df.loc[:, ("Pregnancies", "Glucose", "BloodPressure", "SkinThickness", "Insulin", "BMI","DiabetesPedigreeFunction", "Age")]y = df.loc[:, 'Class']Data = np.array(X)Label = np.array(y)Label = Label * 2 - 1return Data, Labelclass SVM:def __init__(self, max_iter=768, kernel='linear'):self.max_iter = max_iterself._kernel = kernel#参数初始化def init_args(self, features, labels):self.m, self.n = features.shapeself.X = featuresself.Y = labelsself.b = 0.0self.alpha = np.ones(self.m)self.computer_product_matrix()#为了加快训练速度创建一个内积矩阵# 松弛变量self.C = 1.0# 将Ei保存在一个列表里self.create_E()#KKT条件判断def judge_KKT(self, i):y_g = self.function_g(i) * self.Y[i]if self.alpha[i] == 0:return y_g >= 1elif 0 < self.alpha[i] < self.C:return y_g == 1else:return y_g <= 1#计算内积矩阵#如果数据量较大,可以使用系数矩阵def computer_product_matrix(self):self.product_matrix = np.zeros((self.m,self.m)).astype(np.float)for i in range(self.m):for j in range(self.m):if self.product_matrix[i][j]==0.0:self.product_matrix[i][j]=self.product_matrix[j][i]= self.kernel(self.X[i], self.X[j])# 核函数def kernel(self, x1, x2):if self._kernel == 'linear':return np.dot(x1,x2)elif self._kernel == 'poly':return (np.dot(x1,x2) + 1) ** 2return 0#将Ei保存在一个列表里def create_E(self):self.E=(np.dot((self.alpha * self.Y),self.product_matrix)+self.b)-self.Y# 预测函数g(x)def function_g(self, i):return self.b+np.dot((self.alpha * self.Y),self.product_matrix[i])#选择变量def select_alpha(self):# 外层循环首先遍历所有满足0= 0:j =np.argmin(self.E)else:j = np.argmax(self.E)return i, j#剪切def clip_alpha(self, _alpha, L, H):if _alpha > H:return Helif _alpha < L:return Lelse:return _alpha#训练函数,使用SMO算法def Train(self, features, labels):self.init_args(features, labels)#SMO算法训练for t in range(self.max_iter):i1, i2 = self.select_alpha()# 边界if self.Y[i1] == self.Y[i2]:L = max(0, self.alpha[i1] + self.alpha[i2] - self.C)H = min(self.C, self.alpha[i1] + self.alpha[i2])else:L = max(0, self.alpha[i2] - self.alpha[i1])H = min(self.C, self.C + self.alpha[i2] - self.alpha[i1])E1 = self.E[i1]E2 = self.E[i2]# eta=K11+K22-2K12eta = self.kernel(self.X[i1], self.X[i1]) + self.kernel(self.X[i2], self.X[i2]) - 2 * self.kernel(self.X[i1], self.X[i2])if eta <= 0:# print('eta <= 0')continuealpha2_new_unc = self.alpha[i2] + self.Y[i2] * (E1 - E2) / eta # 此处有修改,根据书上应该是E1 - E2,书上130-131页alpha2_new = self.clip_alpha(alpha2_new_unc, L, H)alpha1_new = self.alpha[i1] + self.Y[i1] * self.Y[i2] * (self.alpha[i2] - alpha2_new)b1_new = -E1 - self.Y[i1] * self.kernel(self.X[i1], self.X[i1]) * (alpha1_new - self.alpha[i1]) - self.Y[i2] * self.kernel(self.X[i2], self.X[i1]) * (alpha2_new - self.alpha[i2]) + self.bb2_new = -E2 - self.Y[i1] * self.kernel(self.X[i1], self.X[i2]) * (alpha1_new - self.alpha[i1]) - self.Y[i2] * self.kernel(self.X[i2], self.X[i2]) * (alpha2_new - self.alpha[i2]) + self.bif 0 < alpha1_new < self.C:b_new = b1_newelif 0 < alpha2_new < self.C:b_new = b2_newelse:# 选择中点b_new = (b1_new + b2_new) / 2# 更新参数self.alpha[i1] = alpha1_newself.alpha[i2] = alpha2_newself.b = b_newself.create_E()#这里与书上不同,,我选择更新全部Edef predict(self, data):r = self.bfor i in range(self.m):r += self.alpha[i] * self.Y[i] * self.kernel(data, self.X[i])return 1 if r > 0 else -1def score(self, X_test, y_test):right_count = 0for i in range(len(X_test)):result = self.predict(X_test[i])if result == y_test[i]:right_count += 1return right_count / len(X_test)if __name__ == '__main__':svm = SVM(max_iter=768)X, y = create_data()X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.25, random_state=100)svm.Train(X_train, y_train)print(svm.score(X_test, y_test))
五:knn
代码:
"""
coding:utf-8
sorting algorithms:knn
@author: Li Sentan
@time:2021.10.27
@file:shiyansecond3.py
"""from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
import pandas as pd
from sklearn.model_selection import train_test_splitdata = pd.read_csv("diabetes2.csv")
y = data['Class']
x = data.drop('Class', axis=1)
x = x.drop('Unnamed: 0',axis=1)# X_train, X_test, y_train, y_test = train_test_split(x, y, test_size=0.25, random_state=100)
# knn = KNeighborsClassifier(n_neighbors=14)
# knn.fit(X_train,y_train)
# y_pre = knn.predict(X_test)
# count1 = 0
# count2 = 0
# j = 0
# for i in y_test:
# if y_pre[j]==i:
# count1 = count1 +1
# else:
# count2 = count2 + 1
# j = j + 1
# print("the accuracy:",count1/len(y_pre))
# # print(y_pre)
# # print(y_test)k_range = range(1, 31)
k_error = []
# 循环,取k=1到k=30,查看误差效果
for k in k_range:knn = KNeighborsClassifier(n_neighbors=k)# cv参数决定数据集划分比例,这里是按照5:1划分训练集和测试集scores = cross_val_score(knn, x, y, cv=5, scoring='accuracy')a = scores.mean()print(a)k_error.append(1 - a)# 画图,x轴为k值,y值为误差值
plt.plot(k_range, k_error)
plt.xlabel('Value of K for KNN')
plt.ylabel('Error')
plt.show()笔者特别倾心于python的其中一个原因就是它是一门很简洁灵活的编程语言,要是调用系统库函数的话基本就没几行代码了,所以在此亦能体现出来。首先,属性x,类y的构建方法和决策树是一样的,接下来:
knn = KNeighborsClassifier(n_neighbors=k) #定义一个KNeighborsClassifier()对象,n_neighbors=k代表选取k个临近值。 scores = cross_val_score(knn, x, y, cv=5, scoring='accuracy') # cv参数决定数据集划分比例,这里是按照4:1划分训练集和测试集,每次循环,scores会得到5个准确率,因为是从五份数据中从中选取一份作为测试集,所以有五种选择。scoring='accuracy'代表评价模型的方法用accuracy,即准确率。可以看出,这行代码蕴含了三步,首先计算测试样本与训练样本之间的距离,其次选出距离最近的k个点,并以这k个点中占比最多的类别作为该测试样本的类别,最后将预测值与测试集中的class进行比较,得到准确率。对每个k值得到的5个准确率进行求平均,得到结果如下:
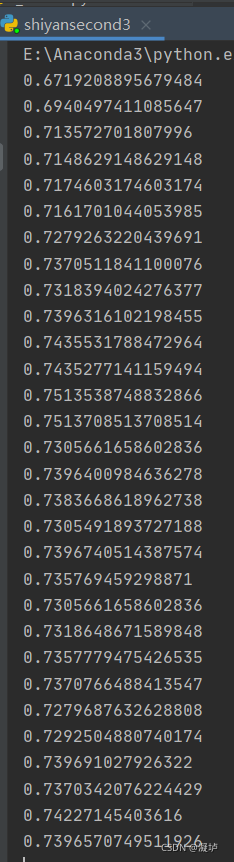
然后对错误率进行分析,做出错误率与k的关系。结果如下:

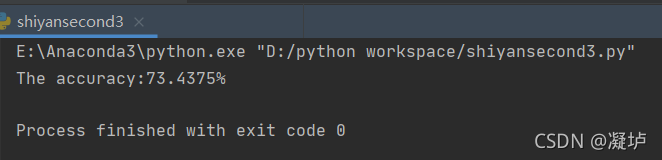
然后,根据图形,我们可以看出,k = 13ork = 14时,错误率是比较低的,所以我们选择这两个k值,手动计算一下准确率,将后面的代码注释掉,中间的代码注释回来即可,中间的代码我用到了fit()函数,下面还有predict(),哈哈哈,完全是一时兴起,但它都没报错,并且predict()返回值是一个class数组,可以看出来,就算knn不用构建模型,但是它跟其他机器学习方法一样能够使用fit(),predict()函数,这不得不令人感慨,python的sklearn库已经做的很成熟了。你只需要在实例化对象时知道函数名称就好啦。
对比之后发现,k=14 的时候准确率比较高:
六:bayes
代码:
"""
coding:utf-8
sorting algorithms:Bayes
@author: Li Sentan
@time:2021.10.28
@file:shiyansecond4.py
"""
import math
import pandas as pd
from sklearn.model_selection import train_test_splitclass Bayes(object):def __init__(self, trainData):self.trainData = trainData# self.inputVector=inputVector# model_par以字典形式存放每一个类别的方差self.model_para = {}count = 0class1 = []for i in trainData:class1.append(i[-1])if i[-1] == 0:count+=1self.a = count/len(trainData)self.b = 1-self.adef tarin_bayesModel(self):# 将训练集按照类别进行提取separated_class = self.separateByClass()# vectors是列表,包含的是每个类别对应的向量集for classValue, vectors in separated_class.items():# 将每一个类别的均值和方差保存在对应的键值对中self.model_para[classValue] = self.summarize(vectors)return self.model_para# 计算均值def mean(self, numbers):return sum(numbers) / float(len(numbers))# 计算方差,注意是分母是n-1def stdev(self, numbers):avg = self.mean(numbers)variance = sum([pow(x - avg, 2) for x in numbers]) / float(len(numbers) - 1)return math.sqrt(variance)# 对每一类样本的每个特征计算均值和方差,结果保存在列表中,依次为第一维特征、第二维特征等...的均值和方差def summarize(self, vectors):# zip利用 * 号操作符,可以将不同元组或者列表压缩为为列表集合。用来提取每类样本下的每一维的特征集合summaries = [(self.mean(attribute), self.stdev(attribute)) for attribute in zip(*vectors)]# 将代表类别的最后一个数据删掉,只保留均值和方差del summaries[-1]del summaries[0]return summaries# 将训练集按照类别进行提取,以字典形式存放,Key为类别,value为列表,列表中包含的是每个类别对应的向量集def separateByClass(self):# 字典用于存放分类后的向量集合separated_class = {}for i in range(len(self.trainData)):vector = self.trainData[i]# vector[-1]为每组数据的类别if (vector[-1] not in separated_class):separated_class[vector[-1]] = []# 将每列数据存放在对应的类别下,列表形式separated_class[vector[-1]].append(vector)return separated_class# 假定服从正态分布,对连续属性计算概率密度函数./>def calProbabilityDensity(self, x, mean, stdev):# x为待分类数据exponent = math.exp(-(math.pow(x - mean, 2) / (2 * math.pow(stdev, 2))))return (1 / (math.sqrt(2 * math.pi) * stdev)) * exponent# 计算待分类数据的联合概率def calClassProbabilities(self, inputVector):# summaries为训练好的贝叶斯模型参数, inputVector为待分类数据(单个)# probabilities用来保存待分类数据对每种类别的联合概率probabilities = {}# classValue为字典的key(类别) ,classSummaries为字典的vlaue(每个类别每维特征的均值和方差),列表形式for classValue, classSummaries in self.model_para.items():probabilities[classValue] = 1# len(classSummaries)表示有多少特征维度for i in range(len(classSummaries)):# mean, stdev分别表示每维特征对应的均值和方差mean, stdev = classSummaries[i]# 提取待分类数据的i维数据值x = inputVector[i+1]# 计算联合概率密度probabilities[classValue] *= self.calProbabilityDensity(x, mean, stdev)if classValue == 0:probabilities[classValue] *= self.aelse:probabilities[classValue] *= self.b# 返回概率最大的类别prediction = max(probabilities, key=probabilities.get)return prediction# 计算分类准确率
def calAccuracy(testData, bayes):correct_nums = 0for i in range(len(testData)):# 逐次计算每一个数据的分类类别if testData[i][-1] == bayes.calClassProbabilities(testData[i]):correct_nums += 1return correct_numsdef main():filename = 'diabetes2.csv'df = pd.DataFrame(pd.read_csv(filename))trainData, testData = train_test_split(df.values, test_size=0.25, random_state= 300)bayes = Bayes(trainData)# model为训练之后的bayes分类器模型的概率参数model = bayes.tarin_bayesModel()print(model)correct_nums = calAccuracy(testData, bayes)print("The accuracy: %f%%" % (correct_nums / len(testData) * 100.0))if __name__ == "__main__":main()
决策树运用分类后熵变小的思想,SVM的思路是尽可能构造出一个超平面,使不同类的数据分开,且不同类数据到该超平面的距离之和尽可能大,knn就是单纯的比较测试点与训练点之间的距离,相比之下,Bayes是一种单纯运用概率的一种方法,其思路我们高中已经接触了很多,并且做了大量的题。
对于思想,笔者用老师的两页ppt来表示,如下:


代码分析: 主要是定义的tarin_bayesModel()方法,返回值是self.model_para,为字典类型,键为Class,值为每个属性的均值,方差,如下所示:
{0.0: [(3.3207547169811322, 3.0610147534363112), (111.28048595238684, 24.20751688299456), (71.22604369297977, 11.63681692187576), (19.752021563342318, 15.386303665820186), (71.07277628032345, 102.6770459761066), (31.045049510197018, 6.6559928665440795), (0.4243099730458222, 0.29865466522797146), (31.528301886792452, 12.031541074281865)], 1.0: [(4.8585365853658535, 3.8277223818131576), (142.5091391490586, 28.964067881757458), (75.00130436229325, 12.382690628770598), (21.746341463414634, 17.396023006752408), (102.22439024390243, 143.71045369157778), (35.497094907777374, 6.922607489869842), (0.5469121951219517, 0.36766818493486086), (36.88780487804878, 10.98180748547412)]}
有了每个属性的均值,方差,我们就可以计算联合概率密度了,所以我们定义了calProbabilityDensity(),calClassProbabilities(),前者计算单个属性的概率密度,后者按类别分别计算联合概率密度,乘以对应的P(class)之后选出最大值对应的Class作为测试数据的预测类别。这句话有点绕,看官可仔细回味。
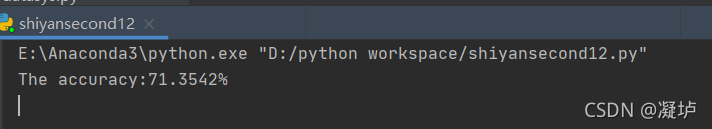
最后调用准确率,判断测试集的本身类别与其对应预测的类别的一致率,结果如下:

笔者发现,其结果与训练集测试集划分的随机数有很大关系,因为bayes本身就是一个概率学思想,其受到数据较大影响是可以理解的。
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!