不好好作图的NCS系列(一):倾斜45°的火山图
数据分析实质和内涵是不会变化的,但是可视化却一直是百花齐放,各有各的样子。我们在读文章的时候,想必也发现过,有些文章在做图上可谓是别出心裁,换着花样。虽然这对于结果没有什么影响,但是在感官上确实更胜一筹。新的一年,我们从火山图开始(祝愿大家红红火火),看看那些不一样的可视化,学习他们的方式方法!
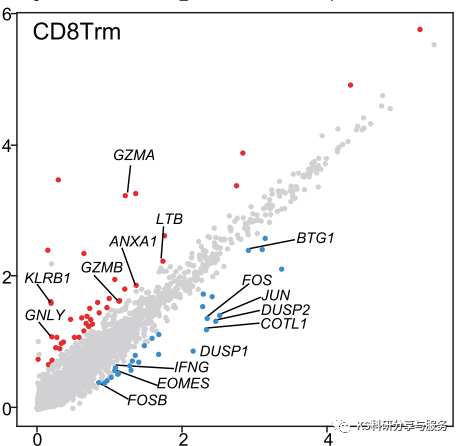

火山图的基本做法我们之前已经讲过了(转录组不求人系列(十): NCS级别的火山图,总有一款适合你!),看看上图这篇Cell文章的火山图,它居然是倾斜的,其实很多NCS的文章在火山图的可视化上都采用这样的模式。接下来我们就学习下如何制作吧!
画这种火山图需要的数据还是差异分析结果,但是需要两列数据,两个分组的平均表达量。之前用DESeq分析差异基因的时候(转录组不求人系列(七):DESeq2分析转录组测序数据),我们在最后得到两个结果,一个是dds1,一个是res,通过这两个数据我们先计算平均值并合并,然后作图。
baseA <- counts(dds1, normalized=T)[, dds1$condition=="Mcc"]if (is.vector(baseA)){baseMeanA <- as.data.frame(baseA)} else {baseMeanA <- as.data.frame(rowMeans(baseA))}colnames(baseMeanA) <- "Mcc"head(baseMeanA)baseB <- counts(dds1, normalized=T)[, dds1$condition=="Pan"]if (is.vector(baseB)){baseMeanB <- as.data.frame(baseB)} else {baseMeanB <- as.data.frame(rowMeans(baseB))}colnames(baseMeanB) <- "Pan"head(baseMeanB)
画图需要使用到ImageGP这个包,先安装加载需要的包:
devtools::install_github("Tong-Chen/ImageGP")library(ImageGP)library(ggplot2)library(ggpubr)library(egg)library(ggrepel)
读取数据,并进行处理和定义:
setwd("E:/生物信息学")diffexpr <- read.csv("df.csv",header = T)diffexpr$Mcc<- log2(diffexpr$Mcc+1)#对平均值进行标准化diffexpr$Pan<- log2(diffexpr$Pan+1)#对平均值进行标准化diffexpr$level <- ifelse(diffexpr$padj<0.05,ifelse(diffexpr$log2FoldChange>=2, "Up",ifelse(diffexpr$log2FoldChange<=-2, "Down", "NoSig")),"NoSig")#标记差异基因head(diffexpr)
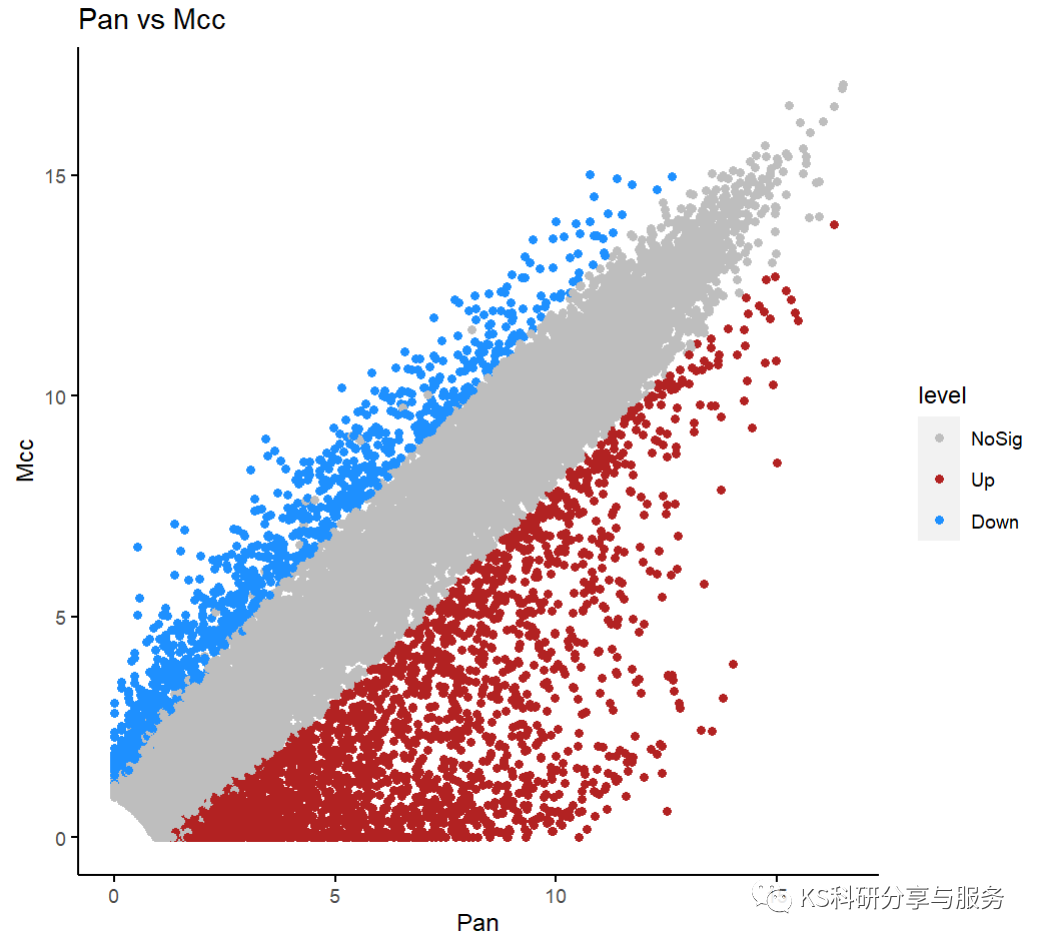
接着就可以画图了:
p <- sp_scatterplot(diffexpr, xvariable = "Pan", yvariable = "Mcc", #定义横纵坐标变量,用的是前面计算的样本平均值color_variable = "level",#颜色以分组定义title ="Pan vs Mcc", #标题color_variable_order = c("NoSig","Up", "Down"),manual_color_vector = c("grey","firebrick","dodgerblue")) + #颜色定义coord_fixed(1)+ labs(x = "Pan", y = "Mcc")p
之后需要标记显著的基因名称,和之前画火山图一样,定义一下:
diffexpr$label =""diffexpr <- diffexpr[order(diffexpr$padj),]up.genes <- head(diffexpr$X[which(diffexpr$level=="Up")],20)down.genes <- head(diffexpr$X[which(diffexpr$level=="Down")],20)top10genes <- c(as.character(up.genes), as.character(down.genes))diffexpr$label[match(top10genes,diffexpr$X)] <- top10genes
之后将基因名称添加至热图即可:
p + geom_text_repel(data=diffexpr, aes(label= label), color="black", size=4, fontface="italic",arrow = arrow(ends="first", length = unit(0.01, "npc")), box.padding = 0.2,point.padding = 0.3, segment.color = 'black', segment.size = 0.3, force = 1,max.iter = 3e3)
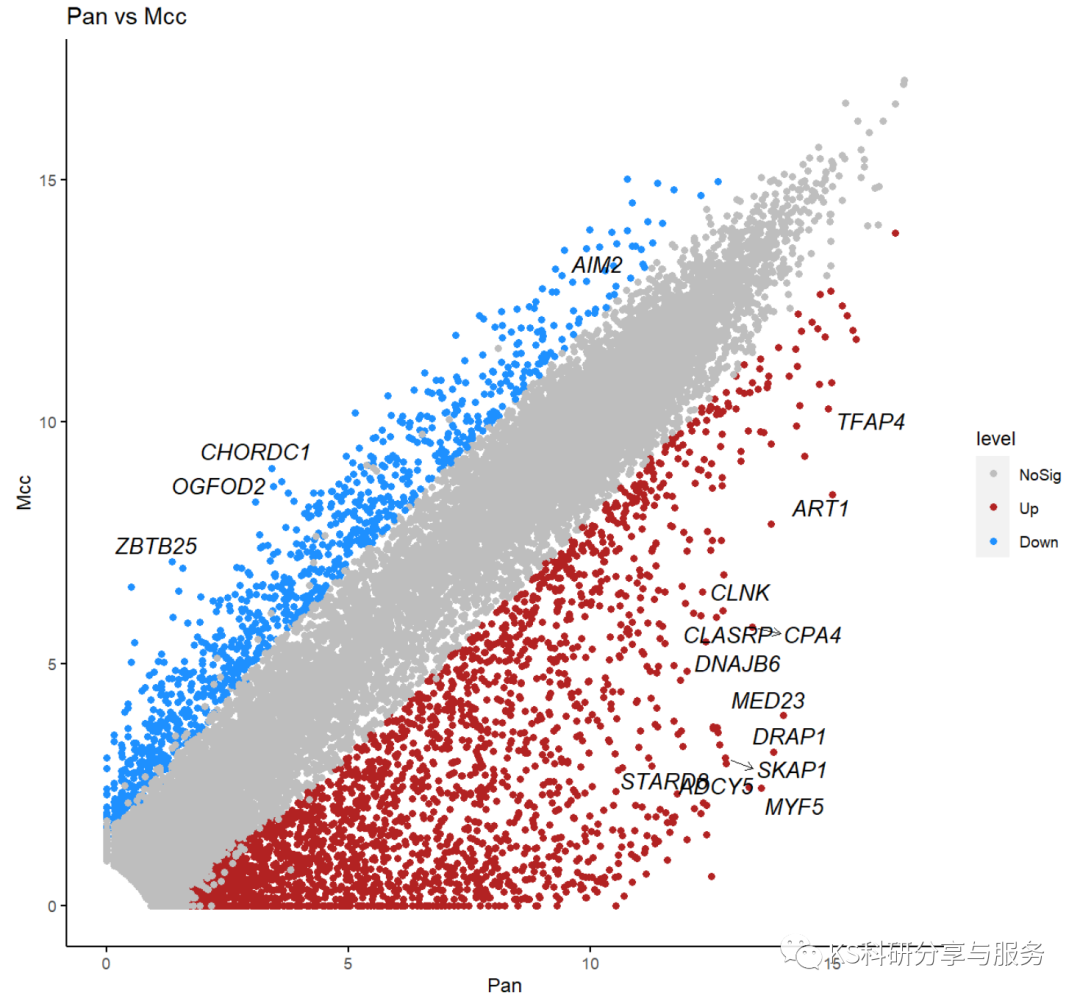
这个图就完成了,如果您也看到一些有意思的图,欢迎私信交流。想要获取示例数据,可打赏截图私信作者获得,记得附上邮箱,感谢支持!
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!