几种国内芯片测序格式和 Affymetrix 位点集格式的对比
前3篇文章都是对比和 Illumina 芯片有关的原始数据格式,这篇则直接比较和 Affymetrix 芯片有关的原始数据格式。
目前国内常见的测序机构里,23魔方主要使用与 Affymetrix 有关的赛默飞世尔定制芯片,据说新版也用了 Illumina 定制芯片,但真实性未知。而其他测序公司比如微基因、各色等,基本都使用或者改用 Illumina 的芯片(微基因的1.0版用 Affymetrix 的定制芯片)。
而国外常见的5家(23andMe、Ancestry、FTDNA、LivingDNA、MyHeritage)测序机构里,LivingDNA 的 V2 版本改用了 Affymetrix 的定制芯片。且 FTDNA 最早期的 hg18 (NCBI36) 坐标版本格式也是 Affymetrix 的,但很快就变更为 Illumina Omni 系列的定制芯片。
而 Affymetrix 的常用官方芯片数据种类不如 Illumina 格式丰富,因此下述的对比图只选取了较有用的几种类型。
1.国内格式转国外与 Affymetrix 相关格式的转化率图(按基因位点位置转化)

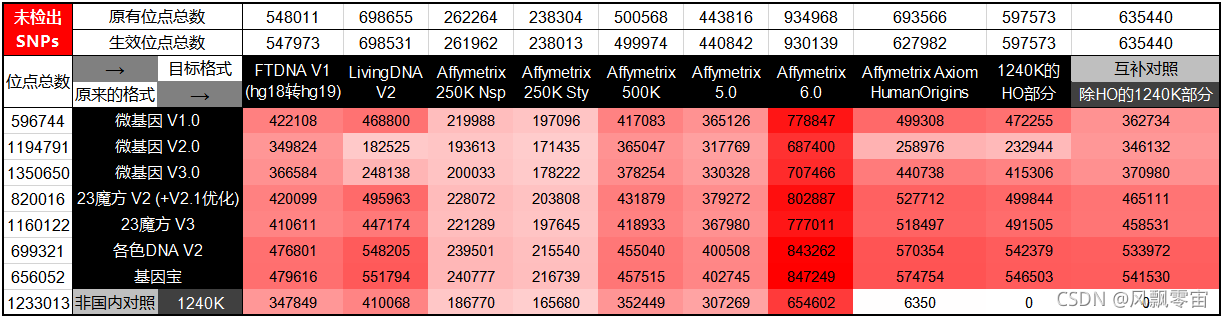
2.国内格式转国外与 Affymetrix 相关格式的未检出位点数图
3.上述格式跑一些祖源计算器的理想利用率图(这里的原始数据文件第4列都填满了纯合位点,其中“[#ANY]”代表这个系列下的任意一个计算器)
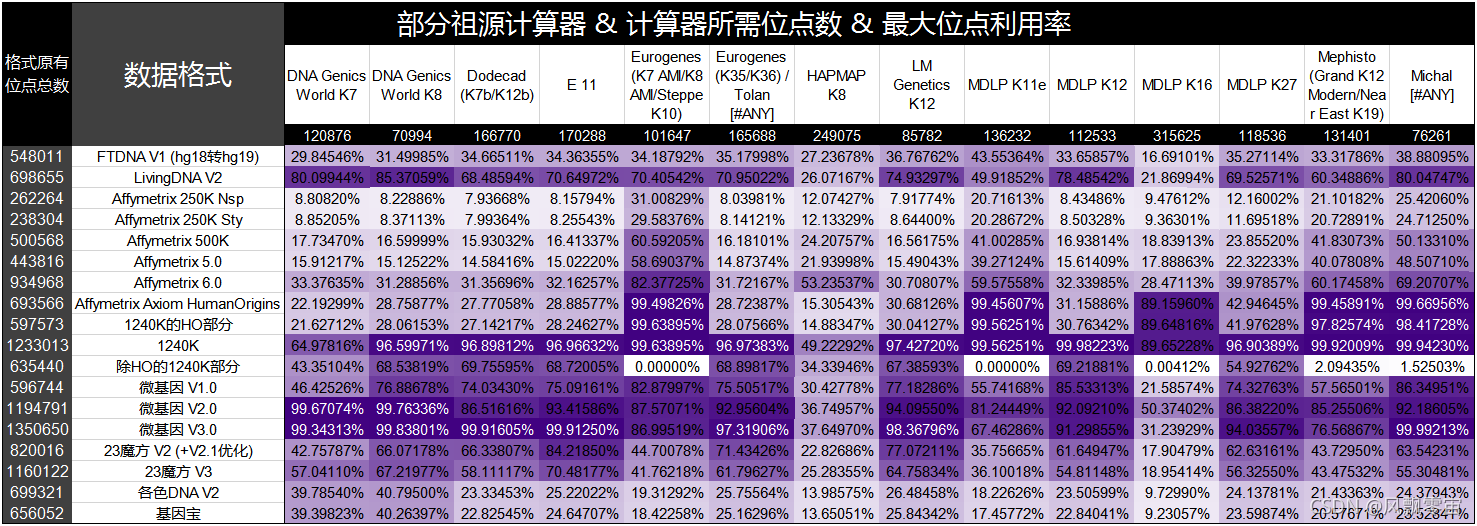
为了方便对比,这次我对 1240K 科研样本格式作了分解,分为 HO(Human Origins,人类起源,部分科研样本是这种格式而非1240K) 部分和非 HO 部分,而且这里用到的 Affymetrix Axiom HumanOrigins 格式位点名以“rs”开头为主,未以大量“Affx-”开头位点为主(在 Axiom 系列格式里,绝大多数基因位点有3种命名方式,少数仅有2种),因此第三幅图中的统计还没达到跑 MDLP K16 这个同时计算“rs”、“Affx-”开头位点计算器的最大位点利用率。
从整体来看,国内格式转化到 Affymetrix 格式的转化率都不理想,仅微基因2.0版对 Affymetrix Human Origins 位点集相对兼容些(同时微基因2.0也更兼容 Illlumina GSA 位点集)。如果您需要用到自己数据的 Affymetrix 官方格式,本人建议使用从全基因组数据的 BAM 或 CRAM 文件提取到芯片格式的方法。
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!