揭秘PLNet:用泊松对数正态图网络分析助力单细胞RNA测序数据处理大突破
今天我们介绍一篇来自北京大学数学科学学院的肖飞轶、唐俊杰发表在NeurIPS 2022会议上的工作,该文章研究了用于计数数据的图形模型估计方法,应用于单细胞基因网络分析。文章介绍了PLN图形模型的概念及其在单细胞基因调控网络分析中的应用。研究表明,该图形模型能够较好地解释单细胞基因表达数据,有助于揭示基因网络的调控机制。文章还探讨了该模型的应用前景和改进方向。

背景介绍
高斯图模型已在许多不同领域广泛应用于直接交互推理。然而,在一些现代应用中,如单细胞RNA测序(scRNA-seq)研究,观察到的数据通常是计数数据,包含许多小计数。传统连续数据图模型不适用于计数数据网络推理。本文研究了泊松对数正态(PLN)图模型——PLNet,适用于计数数据,其中潜在正态分布的精度矩阵表示网络。我们提出了一种两步法估计精度矩阵:首先利用最大边际似然估计器(MMLE)估计潜在协方差矩阵,然后通过最小化套索惩罚d-trace损失函数估计精度矩阵。我们建立了MMLE协方差矩阵的收敛速度,并进一步建立了所提出的高维环境下PLNet精度矩阵估计的收敛速度和符号一致性。尽管PLN模型非亚高斯,但即使随着样本量增加,模型维数指数增长至无穷大,PLNet估计仍是一致的。通过模拟真实scRNA-seq数据和基因调控网络分析,我们评估了PLNet性能,并与现有方法进行了比较。
本文的创新与贡献:
-
本文提出了一种针对计数数据的图模型估计方法,并将其应用于单细胞基因网络的建模。
-
本文提出了一种基于Copula理论的半参数方法,有效地解决了传统的图模型估计方法难以处理计数数据的特殊性质这一问题。
-
本文提出的方法在实际数据集上得到了验证,并取得了比现有方法更好的效果,为相关领域的研究提供了新思路和方法。
方法介绍
本文提出的两步法分为两步:
-
使用最大边际似然估计器(MMLE)估计潜变量协方差矩阵。具体来说,对于潜变量的每个维度,通过最大化其边际对数似然函数来估计其均值和方差。然后将这些均值和方差与协方差矩阵组合起来估计整个潜变量协方差矩阵。这个过程可以通过牛顿-拉夫逊算法来实现。此外,还可以通过一些方法来减小高维度估计中的数值积分计算。最后,对得到的协方差矩阵估计器进行投影操作,使其成为半正定矩阵,以保证在D-trace方法中使用时的凸性。
-
通过最小化带套索惩罚的D-trace损失函数来估计精度矩阵。
除此之外,作者证明了在高维设置下,MMLE的协方差矩阵估计的收敛速度,进一步证明了该方法估计精度矩阵的收敛速度和符号一致性。
实验介绍
数据集:本文使用了多组模拟数据集和真实的单细胞转录组数据集。模拟数据集是通过基于图形模型的方法生成的,其中数据维度从100到500不等,样本大小为100到200个。而真实数据集是从“大脑单细胞转录组”和“脂肪细胞单细胞转录组”中获得的,分别包括不同细胞类型的单细胞RNA测序数据。其中,“大脑单细胞转录组”包括来自小鼠额叶、海马、纹状体等部位的单细胞RNA测序数据,而“脂肪细胞单细胞转录组”则包括人类皮下脂肪细胞的数据。这些数据集都是公开可用的,作者提供了相关数据的链接和详细描述。同时,作者还对模拟数据集和真实数据集进行了不同程度的随机采样和过滤等处理,以验证算法的鲁棒性和效果。
实验过程:本文主要介绍了一种基于图模型的计数数据建模方法,并将其应用于单细胞基因网络的估计。实验部分包括两个方面:一是通过模拟数据进行了性能评估,二是利用真实的单细胞转录组数据进行了应用实例的展示。
对于模拟数据的实验,本文设置了四种不同的图结构,并根据这些结构生成数据集进行实验。在每个图结构下,本文分别生成了100个数据集进行评估,用平均绝对误差和平均相对误差来评价模型的性能。
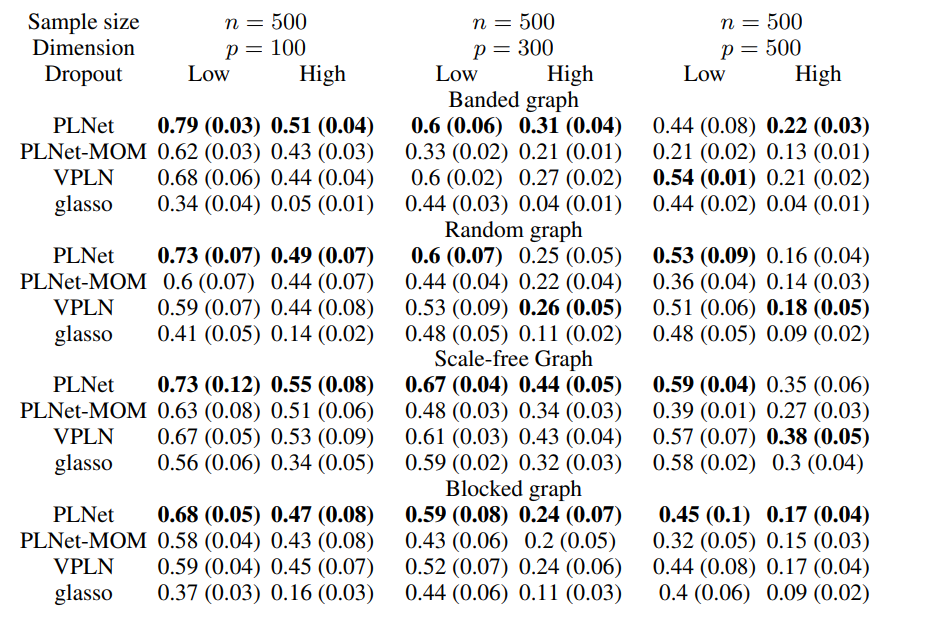
表1:PLNet, VPLN, glasso和PLNet - MOM在n = 500仿真结果的精度下面积和召回曲线(AUPR)方面的比较。结果为超过100次重复的平均值,括号内为标准差。
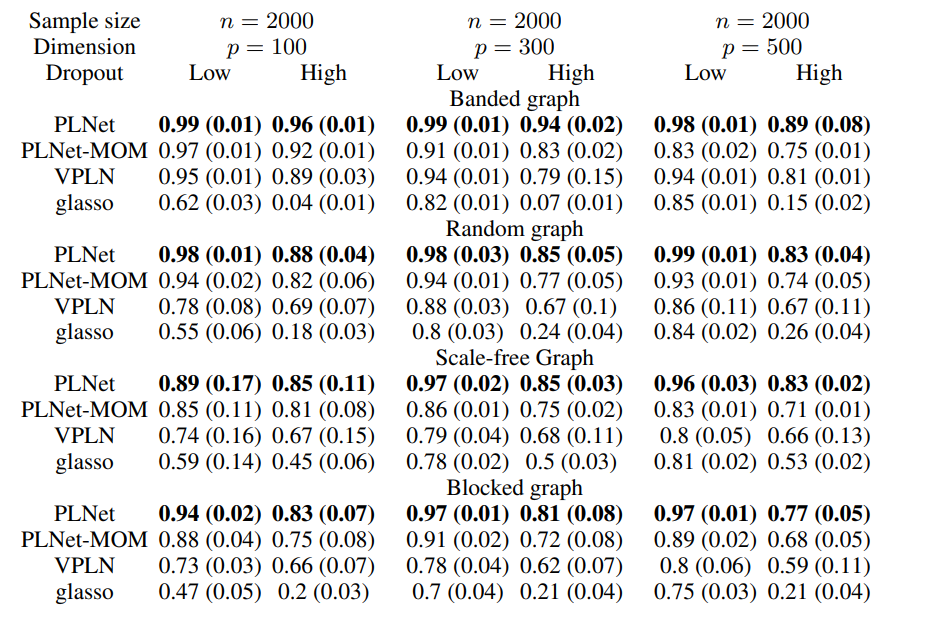
表2:在n = 2000的模拟结果上,PLNet、VPLN、glasso和PLNet - MOM在精度下面积和召回曲线(AUPR)方面的比较。结果为超过100次重复的平均值,括号内为标准差。
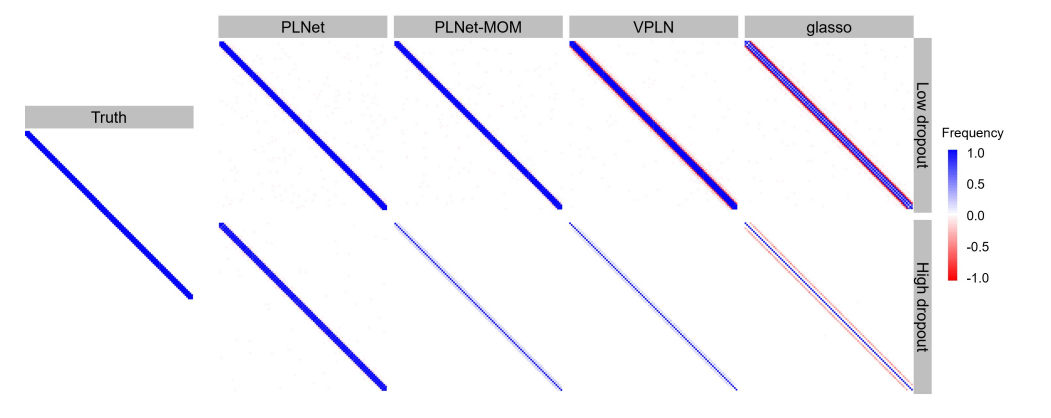
图1:用PLNet、VPLN、glasso和PLNet - MOM对n = 2000的100个节点带状图预测的平均网络。假边用红色标注,真边用蓝色标注。左面板为真实网络矩阵,供参考。
在应用实例方面,本文选取了一个真实的单细胞转录组数据集,并按照本文提出的方法进行建模和估计。
实验结果:本文的实验结果主要涉及不同方法在模拟数据和真实数据上的表现。在模拟数据中,本文比较了PLNet、VPLN、glasso和PLNet-MOM这四种方法在不同数据维度和不同dropout水平下的表现,主要通过计算面积下的精确度和召回率曲线(AUPR)来进行评估。结果表明,PLNet在低和高dropout水平下均比其他三种方法表现更好。在真实数据中,本文将PLNet和VPLN应用于单细胞基因网络分析中,比较它们在DetectingEMT和MouseBrain数据集上的表现。实验结果表明,PLNet成功识别了已知的基因调控网络,并且能够发现新的潜在调控关系。此外,本文还可视化了通过四种方法在banded图上预测的平均网络,结果表明PLNet在发现真实网络拓扑结构方面表现优秀。综上,本文的实验结果表明,PLNet在探索单细胞基因网络方面有着很高的表现。
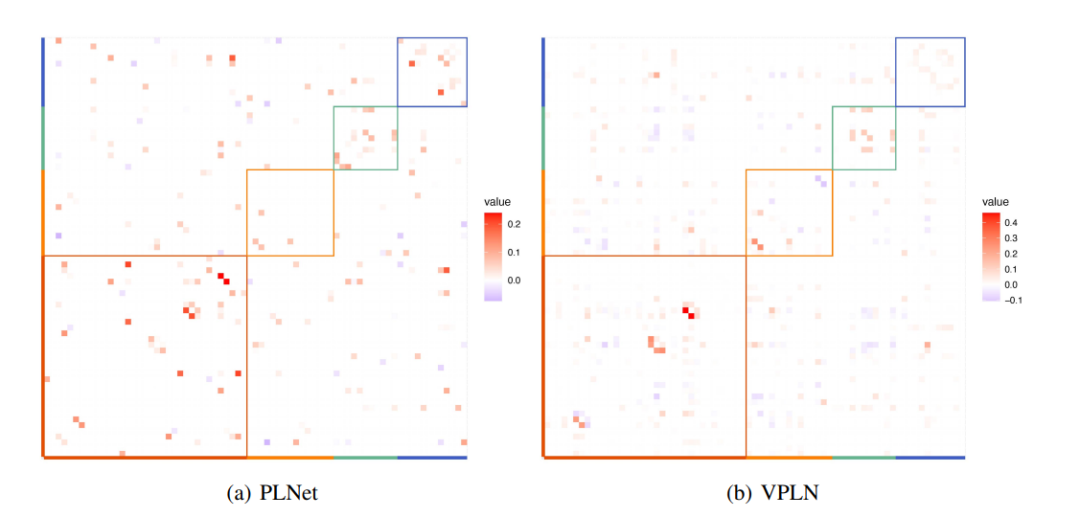
图2:PLNet (a)和VPLN (b)给出的4个GO模块中基因的部分相关性热图。红色:细胞因子介导的信号通路(模块M1);橙色:中性粒细胞介导免疫(模块M2);绿色:细胞蛋白质代谢过程(模块M3);蓝色:蛋白水解(模块M4)
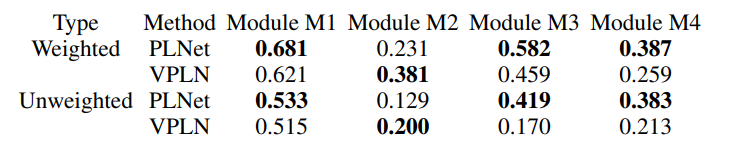
表3:将PLNet和VPLN估计的网络中4个模块的内间连接率调优,使网络密度在5%左右

表4:两种方法在不同密度水平下估计的真边数
结果与讨论
本文考虑了计数数据的PLN图形模型。该模型对单细胞基因调控网络分析有较直观的解释。泊松层用于捕捉9种技术噪声,对数正态层用于模拟单细胞的生物波动。基因调控网络用潜对数正态模型的精度矩阵表示。为了估计精度矩阵,本文提出了一个两步估计器PLNet,使用MMLE估计协方差矩阵,然后最小化惩罚d-trace损失来估计精度矩阵。这种估计过程的简单性使本文能够为所提出的PLNet估计器建立高维设置的一致性理论。数值分析也表明,PLNet方法优于现有方法。
参考资料
Xiao, Feiyi, Junjie Tang, Huaying Fang, and Ruibin Xi. "Estimating graphical models for count data with applications to single-cell gene network." Advances in Neural Information Processing Systems 35 (2022): 29038-29050.
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!