复现Nature medicine图表---堆叠柱状图显示每个样本上下调差异基因
学习单细胞文章的时候,看到NM上一篇文章差异基因的显示方法。

图形如下:

先解释下这样展示的意义(举例可能并不是很恰当):假设我们需要研究某个细胞群分为两个亚群,想要看这两个亚群之间的差异基因变化情况,就可以使用这种方法。先计算每个样本中这两群细胞的差异基因,再将所有样本合并,找出需要关注的基因,就可以作图了。
因为我们演示数据并没有涉及这样的意义,所以偷懒用了不同群细胞的差异基因构建了用来演示的示例数据。图形本身是柱状图,而且是堆叠柱状图。那么作图需要以下因素,首先是纵坐标的LogFC,横坐标的基因名,还有分组。具体的数据形式如下(差异基因筛选不再赘述):
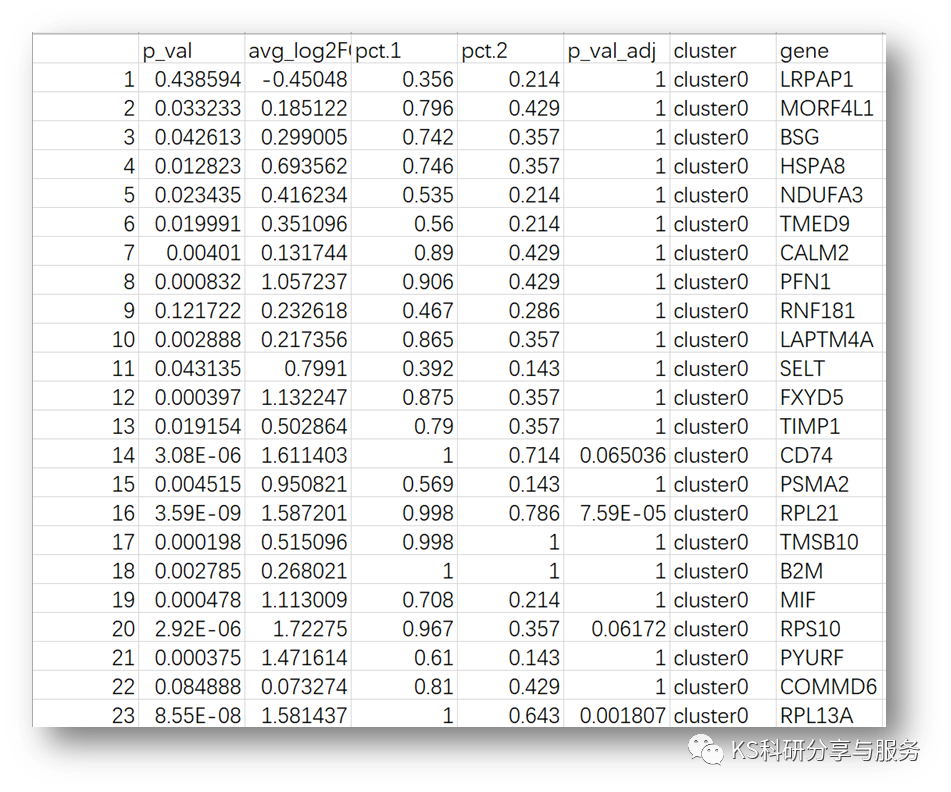
接下来用ggplot作图即可,为了让基因排列按照我们拍好的顺序,将其转化为因子排序,这样作图显示基因名顺序就不会变了。
diff <- read.csv("diff.csv",header = T,row.names = 1)
library(ggplot2)
library(forcats)
diff$gene <- as.factor(diff$gene )
diff$gene <- fct_inorder(diff$gene
先做一个基本的柱状图。
ggplot()+geom_bar(data=diff,aes(x=avg_log2FC,y=gene,fill=cluster),stat="identity",colour = "grey50")#柱子边框颜色

接下来,改变主题,图形方向,填充颜色即可。
ggplot()+geom_bar(data=diff,aes(x=avg_log2FC,y=gene,fill=cluster),stat="identity",colour = "grey50")+scale_fill_manual(values = c("#2271b6","#6bafd6","#9ecbe2","#fd9272","#fee1d3"))+theme_bw()+labs(x="Cumulative avg. logFC",y="Gene expression")+coord_flip()+theme(axis.text.x=element_text(angle=90,hjust = 1,vjust=0.5))#X轴
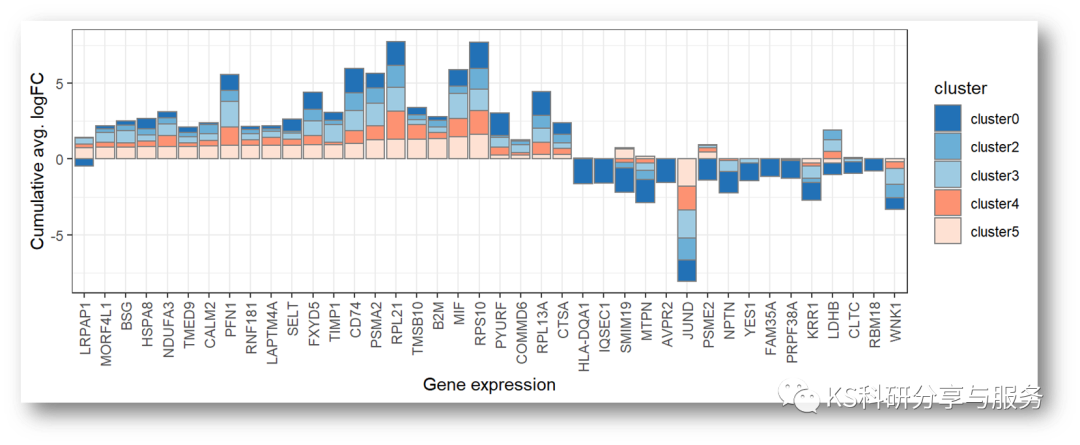
这样就完成了,就是一摸一样有没有?解锁新的数据展示,快尝试下吧。想要示例数据的小伙伴可以到我的公众号《KS科研分享与服务》,感谢支持!
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!