Design the Rac/Raf interface小菜鸟中文教程
关键词:设计界面
本文是根据 /*(rosetta路径)/demos/public/design_raf_rac_interface 教程写的自己实际运行遇到的问题和解决办法。作为笔记~
用途
导师让我用这个小程序设计蛋白蛋白相互作用界面,以增强它们的亲和力。
步骤
1.准备好docking完以后待改进亲和力的复合物pdb文件,如prepacked_0998.pdb 。有A链和B链,其中B链是我想突变的链。
2.准备好design_script.xml文件,在/*(rosetta路径)/demos/public/design_raf_rac_interface/rosetta_inputs 文件夹中。
可能需要修改的地方:
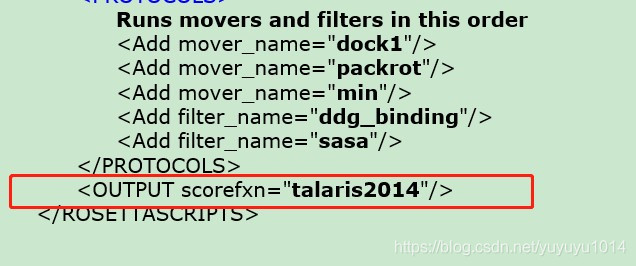
在xml文件最后加入一句OUT PUT scorefxn=“talaris2014”/ ,如图,记得加<> 。
还有一个地方,可以先不用管,但是如果突变完发现突变错链了,再修改就行。

第一行:等号后边的数字,”0”代表no , “1”代表yes。不用设计链2,设计链1。需要重装链2,需要重装链1。(repack怎么翻译?)
第二行:链1需要设计和重装表面残基,并且链2只需要重装。
我针对自己的改成了这样:repack_chain1=“0” repack_chain2=“1” design_chain1=“0” design_chain2=“1”
Designs and repacks interface residues of chain 2 and repacks interface residues of chain 1
3.输入命令:rosetta_scripts.static.linuxgccrelease -s prepacked_0998.pdb -parser:protocol design_script.xml -in:file:native prepacked_0998.pdb -ex1 -ex2 -ignore_unrecognized_res -restore_talaris_behavior -nstruct 1 -overwrite
README里面是没有-restore_talaris_behavior这个命令的,但是我运行的时候报错了!加上能解决。
-nstruct 的值先设置为1.运行成功以后再设置为1000。
运行完也许会得不到结构,出现报错。如图。
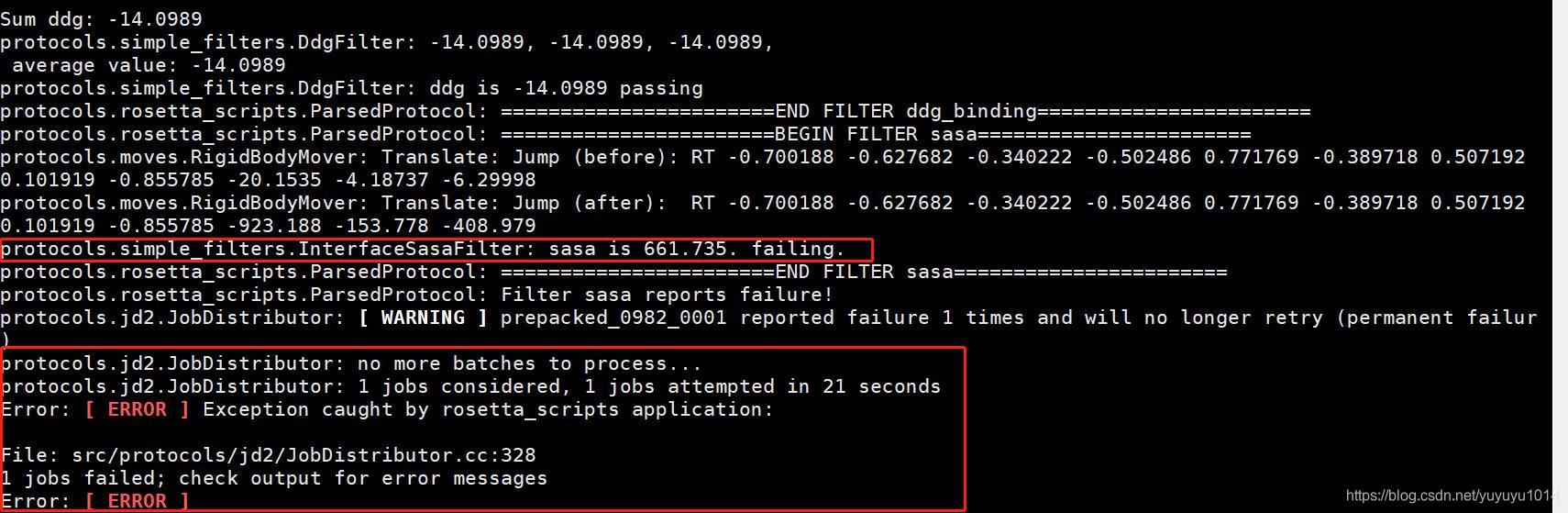
这里是因为sasa的值(661.375)小于了脚本里面设置的800。可以多试几次,实在不行就换个结构突变吧。也是可能因为这个原因,设置输出1000个,有时候得不到1000个。
4.上面步骤得到了1000个突变结构,根据score找到排名靠前的结构进行分析康康。
使用小插件SequenceProfile.py 具体位置
/*/tools/protein_tools/scripts

- :首先它不能处理复合链只能处理单练,根据README里的命令把突变的B链分出来。
grep " B " prepacked_0998.pdb > chainBin.pdb
grep " B " prepacked_0998_555.pdb > chainBin_555.pdb (需要对比的突变结构,这里是根据score排名最好的结构) - ls chainBin_555.pdb > list.txt
把要分析的结构存在 list.txt文件中 - 运行命令
python2 /*/tools/protein_tools/scripts -l list.txt -t chainBin.pdb
就能看到是哪些残基突变了!
还可以继续用analyzing_interface_quality 来分析界面,教程知乎上面有,rosetta研习社的。
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!