实验篇——ks密度分布图的绘制
实验篇——ks密度分布图的绘制
文章目录
- 前言
- 一、ks、ka 介绍
- 1. ka 介绍
- 2. ks 介绍
- 3. ka/ks 介绍
- 二、Ks密度分布图
- 三、代码实现
- 总结
前言
在得到一个全基因组复制(WGD)分析结果文件后,如何对数据集中的ks值进行ks密度分布图的绘制。本章就简单介绍一下方法。
一、ks、ka 介绍
1. ka 介绍
在生物信息学分析中,Ka是一种表示非同义替换的指标。Ka是一个衡量遗传变异(突变)在基因组中的速率的指标。它代表每个位点上非同义替换突变的速率,也就是遗传编码中由于突变而导致氨基酸替换的频率。
非同义替换是指在基因的DNA序列中,单个核苷酸发生突变从而导致编码的氨基酸发生改变的现象。非同义替换是产生SNP位点的一种常见机制。
2. ks 介绍
ks是一个表示同义替换的指标。与Ka相对应,ks代表每个位点上发生同义替换的速率,即由于突变而不改变氨基酸的频率
同义替换是指在基因的DNA序列中,单个核苷酸发生突变,但由于遗传密码子的特性,编码的氨基酸序列并未改变的现象。
3. ka/ks 介绍
Ka/Ks是一种常用于基因比较和选择压力分析的统计指标,它用于衡量非同义替换(Ka)和同义替换(Ks)的相对速率。
当Ka/Ks > 1时,意味着非同义替换的速率高于同义替换,暗示该基因可能受到正向选择推动。这可能表示蛋白质功能的重要改变或对环境适应的需要。相反,当Ka/Ks < 1时,同义替换的速率高于非同义替换,这可能表示该基因受到较强的保守或中性选择。
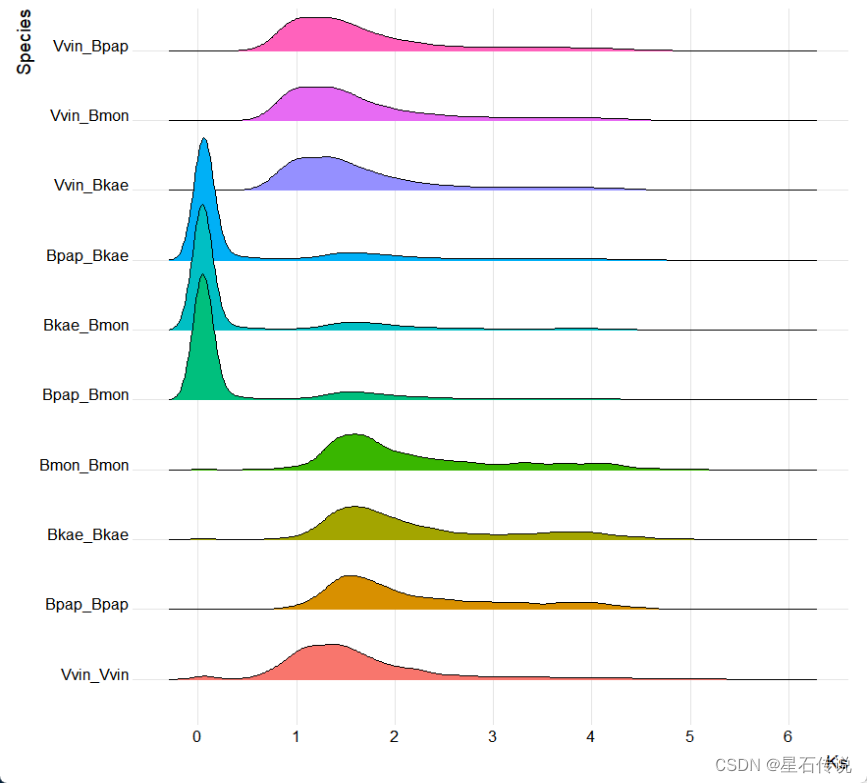
二、Ks密度分布图
通过绘制Ks密度分布图,可以帮助我们观察和分析基因复制事件的累积情况以及基因家族的演化过程,
-
可用于比较不同物种或不同基因组之间的基因复制事件和进化速率。通过观察Ks密度分布图的差异,可以揭示物种间、群体间或基因组间的进化过程和演化特征。
-
展示在不同Ks值范围内的基因数量。如果在较小的Ks值范围内有相对较多的基因,说明基因复制事件在物种分化后的短时间内较为活跃,反之则说明较早期的基因复制事件较为频繁。
-
可以提供基因复制事件的分布情况。如果Ks密度分布图呈现峰值或者非单峰的分布,可以推断出不同时期发生的基因复制事件的存在。特别是当出现多个峰值时,可能表示不同的基因复制事件在不同的时间点发生。
-
可以推测不同基因复制事件的进化速率。较小的Ks值峰值和高峰值区间可能表示较为近期和较快速的基因复制事件。而较大的Ks值峰值和高峰值区间则可能表示较早期和较慢速的基因复制事件。
三、代码实现
在R语言中使用ggplot2包以及其扩展包ggridges,来实现ks密度分布图的绘制
#读取存有KS值的文件
wenjian <- read.table("D:/goushu.ks.txt",header=TRUE,encoding = "utf-8",sep="\t")#查看文件的数据信息
dim(wenjian)
table(wenjian$Species)
class(wenjian$Species)
> tail(wenjian)gene Ks Ka Ka.Ks Species
121436 BkaeChr1G00012590.1-BpapChr1G00016350.1 1.9378 1.6476 0.8502 Bpap_Bkae
121437 BpapChr3G00116140.1-rna-XM_019222922.1 2.2379 2.0686 0.9244 Vvin_Bpap
121438 BpapChr13G00087970.1-rna-XM_019217106.1 3.9383 2.4206 0.6146 Vvin_Bpap
121439 brm_Hic_asm_12.1208-rna-XM_019217106.1 1.8901 3.2662 1.7280 Vvin_Bmon
121440 brm_Hic_asm_11.275-rna-XM_002278801.4 1.3242 5.1765 3.9091 Vvin_Bmon
121441 BkaeChr3G00119950.1-rna-XM_019222922.1 1.9975 5.8123 2.9097 Vvin_Bkae#将Species列转化为因子,作为分类变量(通过对levels参数的设置,可自行定义纵坐标的排序)
wenjian$Species <- factor(wenjian$Species, levels = rev(c("Vvin_Bpap","Vvin_Bmon","Vvin_Bkae","Bpap_Bkae","Bkae_Bmon","Bpap_Bmon","Bmon_Bmon","Bkae_Bkae","Bpap_Bpap","Vvin_Vvin")))#加载包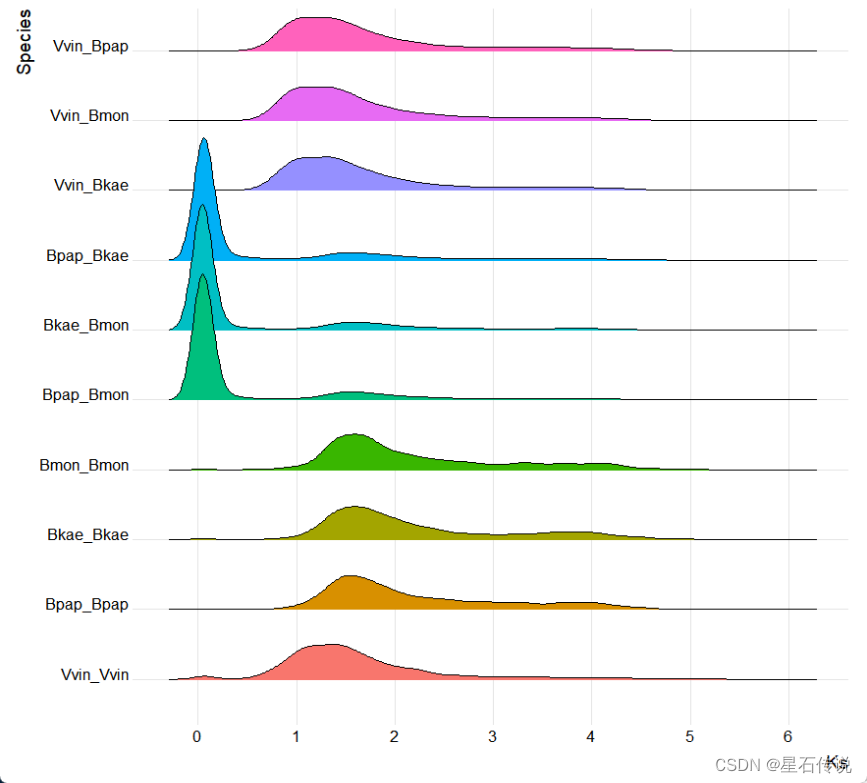library(ggridges)
library(ggplot2)
#install.packages("ggridges")
head(wenjian)#图形绘制
gg <- ggplot(wenjian, aes(y = Species, x = Ks, fill = Species)) +geom_density_ridges_gradient() +theme_ridges() +labs(x = "Ks", y = "Species") +theme(legend.position = "None")+scale_x_continuous(breaks = seq(0,6,by=1))# 指定保存路径和文件名,将图形保存为PNG格式(可自行调整图像的宽度、高度以及像素)
ggsave("D:/ksimage.png",plot = gg)示例图如下:
总结
本章简单介绍了一下Ks、Ka的概念,以及如何用R语言实现Ks密度分布图的绘制。
恰同学少年,风华正茂;
–2023-8-3 实验篇
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!