【GEO Database - 2】sva-ComBat函数去除批次效应
文章目录
- 一、什么是批次效应(batch effect)
- 二、为什么要去除批次效应?
- 三、处理过程
- 1、环境搭建
- 2、读入已标准化过的数据
- 3、对三套数据取交集进行合并
- 4、用Combat()进行处理
- 四、批次处理前后对比
- 1、Heat Map
- 2、PCA
一、什么是批次效应(batch effect)
芯片批次效应是在处理过程中由于技术原因(非生物因素)而添加到样本中的变异。
1、包括使用的扩增试剂的批次,测定完成的时间,甚至大气臭氧水平。如样本制备中的系统变化,扫描仪的差异。
2、还有就是用道不同平台(Illumina/affymetrix)的芯片数据做分析的时候。
二、为什么要去除批次效应?
其他潜在的批次效应在长期研究和meta分析中往往是不可避免的。
标准化虽然可以调整各个样本的测量的全局属性,从而可以更加适当地进行比较。但是,标准化不会消除批次效应。在某些情况下,这些标准化程序甚至可能在高通量测量中加剧技术人为因素。
所以,在处理不同批次的样本时我们需要去除批次效应。
三、处理过程
1、环境搭建
setwd("C:/Users/Administrator/Desktop/lab4-combat-PCA-st/data")
if (!requireNamespace("BiocManager", quietly = TRUE))install.packages("BiocManager")BiocManager::install("sva")
library("sva")
2、读入已标准化过的数据
标准化处理详见用Bionconductor的affy包处理.cel文件
GSE32676 <- read.table("GSE32676_rma_symbol.txt",header=T,row.names=1,sep="\t")
GSE41368 <- read.table("GSE41368_rma_symbol.txt",header=T,row.names=1,sep="\t")
GSE43795 <- read.table("GSE43795_rma_symbol.txt",header=T,row.names=1,sep="\t")
3、对三套数据取交集进行合并
##intersect函数取交集
mrna_names <- intersect(rownames(GSE32676),rownames(GSE41368))
mrna_intersect <- intersect(mrna_names,rownames(GSE43795))
##cbind进行合并
expr <- cbind(GSE32676[mrna_intersect,],GSE41368[mrna_intersect,],GSE43795[mrna_intersect,])
write.table(expr,"../result/mrna_nocombat.txt")
##32,12,11分别是提取出的每个样本的数量,样本1命名为batch1,样本2命名为batch2,样本3命名为batch3
batch <- paste0("batch",rep(c(1,2,3),c(32,12,11)))
##25,7代表样本1中有25个实验组,7个对照组,以此类推
tissue <- rep(c("case","control","case","control","case","control"),c(25,7,6,6,6,5))
table(batch,tissue)
mod <- model.matrix(~tissue)
4、用Combat()进行处理
ComBat是基于经典贝叶斯的分析方法,运用已知的批次信息对高通量数据进行批次校正。
函数输入数据为经过标准化的数据矩阵,返回结果为经过批次校正后的一个数据矩阵
expr_batch<-ComBat(dat=expr,batch=batch,mod=mod)
write.table(expr_batch,"../result/mrna_expr_batch.txt")
write.table(expr_batch[,1:32],"../result/GSE32676_after_batch.txt")
write.table(expr_batch[,33:44],"../result/GSE41368_after_batch.txt")
write.table(expr_batch[,45:55],"../result/GSE43795_after_batch.txt")
四、批次处理前后对比
1、Heat Map
install.packages("gplots")
library(gplots)
heatmap.2(as.matrix(expr),cexrow=0.8,cexcol=1.0)
heatmap.2(as.matrix(expr_batch),cexrow=0.8,cexcol=1.0)
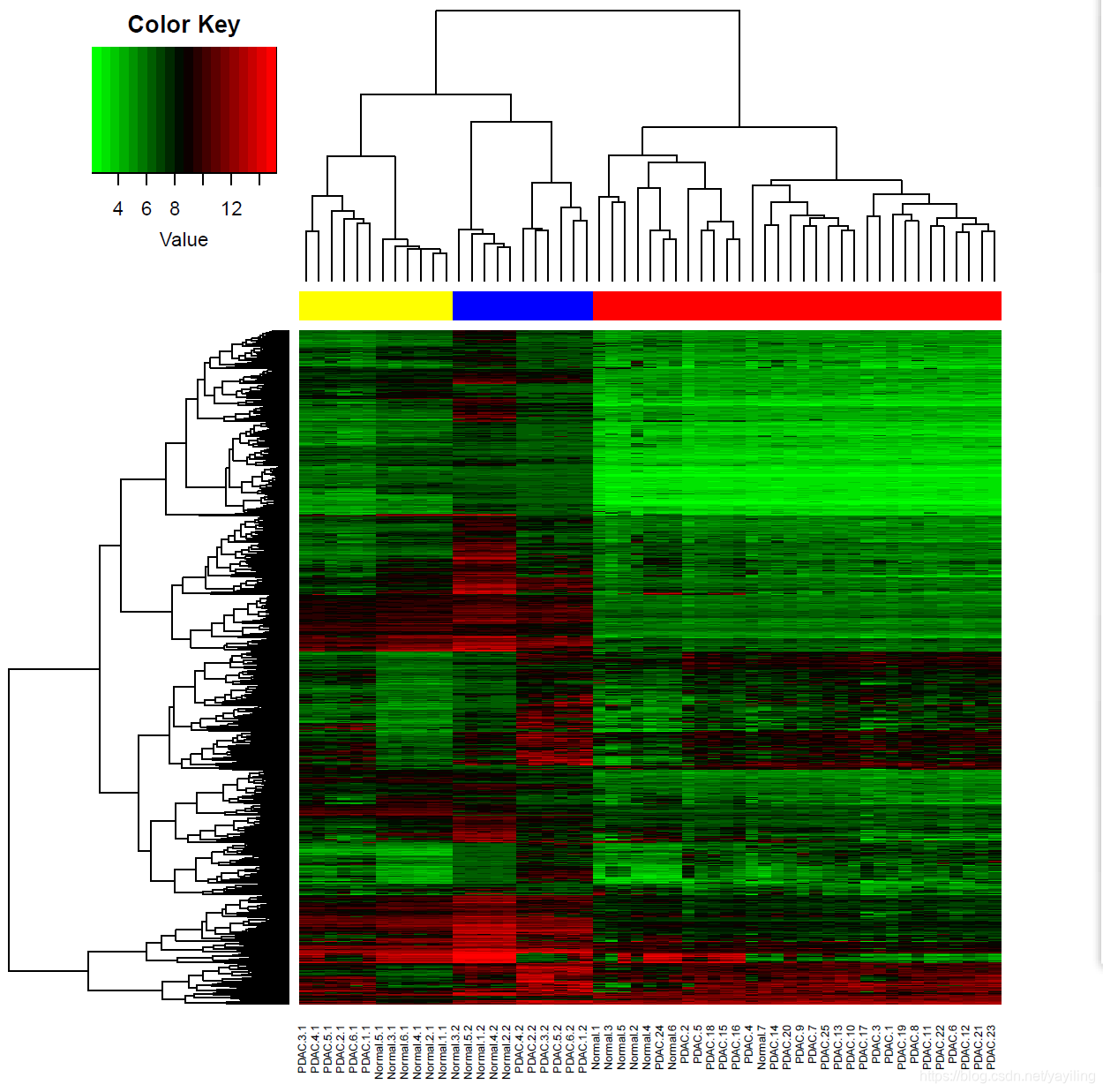
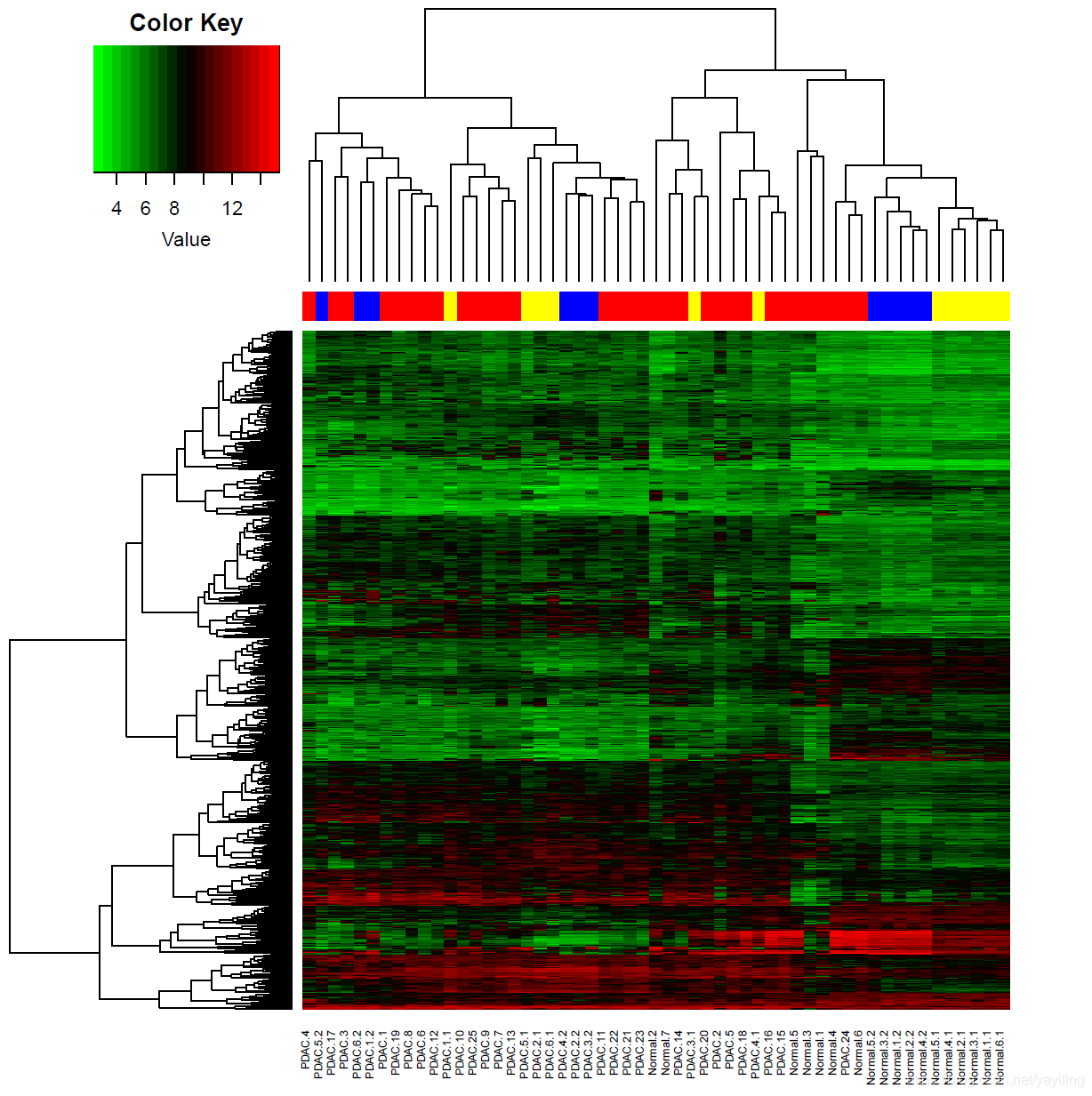
可以观察到之前是按照批次聚类,现在批次差异消除了
2、PCA
pca.plot(expr)
pca.plot(expr_batch)
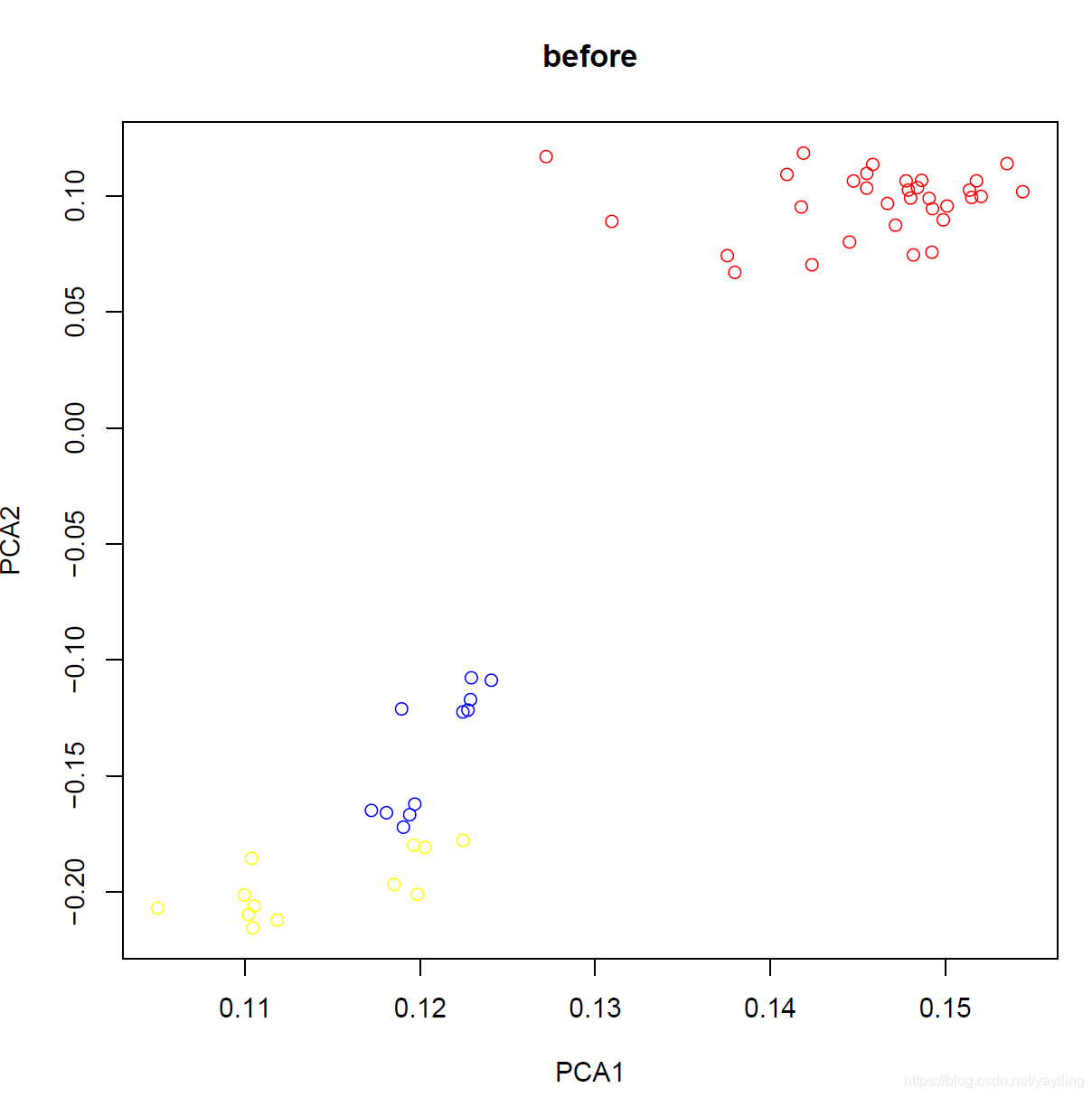
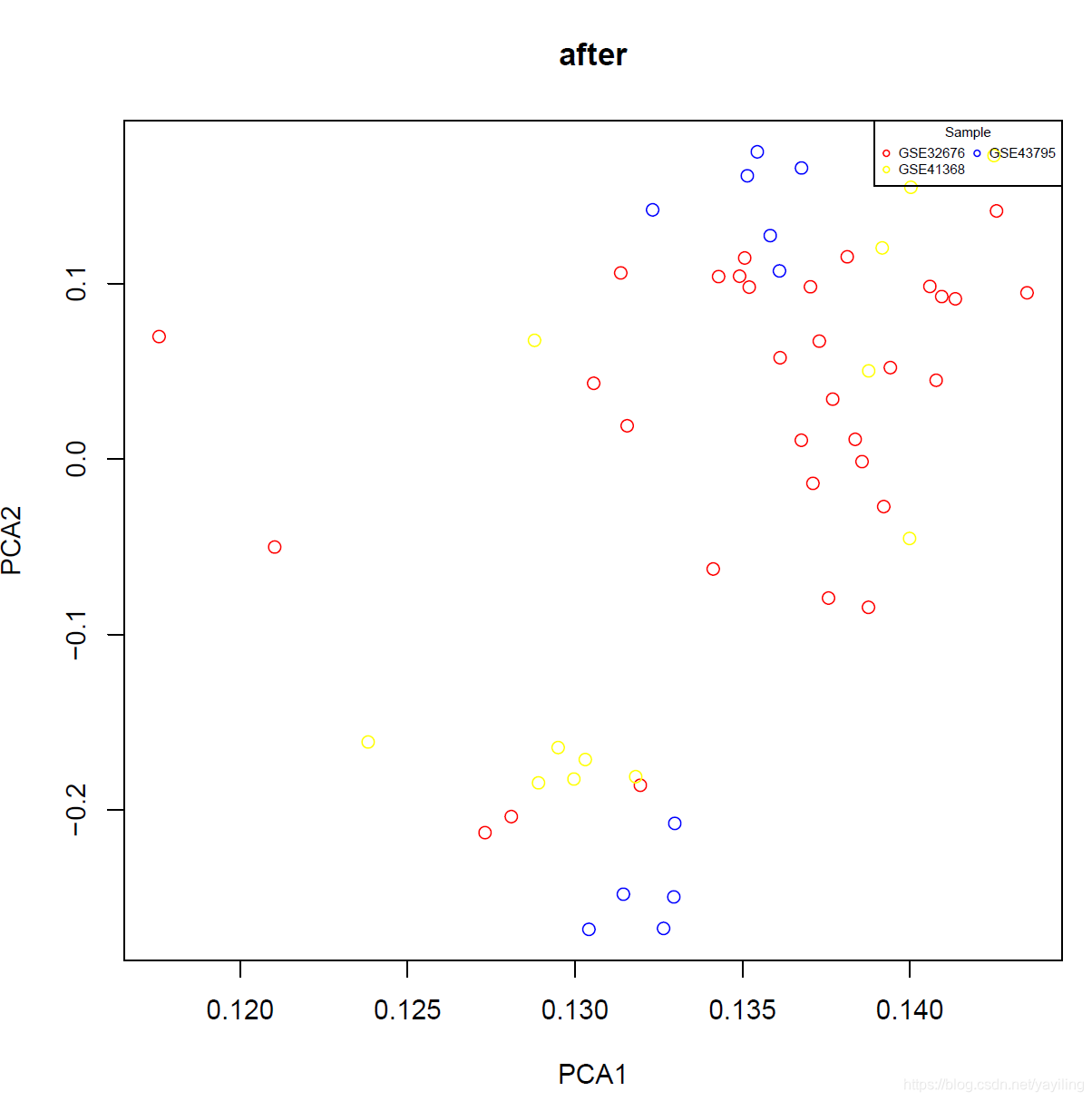
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!