Nucleic Acids Res. | PASSer: 快速准确预测蛋白质变构位点
变构调控是蛋白质活性调控中关键的生物学过程。变构过程指的是蛋白质与小分子结合改变蛋白质的构象,导致其生物活性改变的现象。变构调控过程的一些特征可以应用于药物设计:(a)变构位点在蛋白质进化中具有高度特异性,(b)变构药物可以激活或抑制蛋白质活性,产生潜在的治疗效果,(c)基于变构位点的治疗效果是可控的。出于这些原因,变构位点的研究对于变构药物的开发至关重要,并在近年中得到广泛重视。

2023年4月27日,美国德州南卫理公会大学陶鹏团队在Nucleic Acids Research上发表了题目为PASSer: fast and accurate prediction of protein allosteric sites的研究论文。该文章针对蛋白质变构位点预测提供了三种机器学习模型,并搭建了蛋白质变构位点网站https://passer.smu.edu。到目前为止,PASSer已被来自70多个国家的研究团队访问超过49,000次,并执行了超过6,200个任务。
机器学习模型
PASSer网站搭载了三种机器学习模型:(1)包括了XGBoost和图卷积神经网络的集成学习模型,(2)自动机器学习(autoML)模型,(3)排序学习(Learning-to-rank)模型。其中,自动机器学习方法基于亚马逊开发的AutoGluon模型。
作者使用了上海交通大学张健老师团队开发的蛋白质变构数据库(Allosteric Database, ASD)以及由A. Zlobin等开发的CASBench数据库,基于FPocket方法检测每一个蛋白质结构的潜在位点,并用19个物理和化学描述符进行位点特征化。作者通过计算每个位点和变构小分子的距离进行自动数据标注,相关代码可通过github下载:https://github.com/smu-tao-group/PASSerRank。
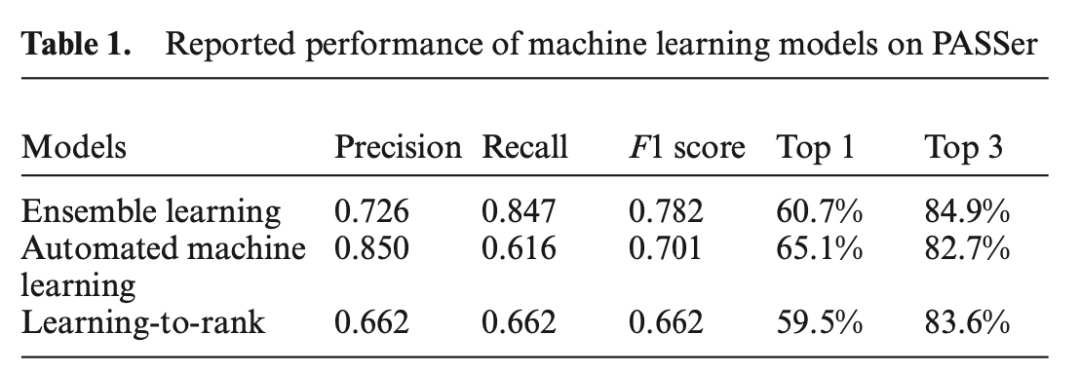
三种机器学习模型性能比较
网页简介

PASSer网站设计结构
此蛋白质变构位点网站可以通过https://passer.smu.edu访问。用户可以输入PDB ID或提交自定义PDB文件。网站默认对体系中所有的蛋白质链进行分析,用户也可以选择特定的链ID。在三种机器学习模型中,集成学习模型和排序学习一般在1-2秒内完成预测任务,自动机器学习模型则需要20秒左右。结果页面列出预测结果前三的位点、概率、以及相关氨基酸,并在可交互界面内展示蛋白质结构和位点位置及形状。
结论
PASSer是一个用于预测蛋白质变构位点的网页应用程序。它提供了三个机器学习模型,以实现可靠和准确的预测性能,并提供了可交互的预测结果。该网站托管在SMU高性能计算平台上,使其能够在几秒内完成预测。PASSer已广泛应用于验证已知功能性位点以及发现新的变构位点。
参考资料
Hao Tian, Sian Xiao, Xi Jiang, Peng Tao, PASSer: fast and accurate prediction of protein allosteric sites, Nucleic Acids Research, 2023;, gkad303,
https://doi.org/10.1093/nar/gkad303
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!