系统发育树的生成与美化(MEGA7和iTOL)--1.MEGA7生成系统发育树
#小白学习生成系统发育树
1.序列准备-本文中的数据LOC号是在文献中的附件中所找到的,然后在Phytozome v13中逐个查找氨基酸序列,需要输入所查找的物种名称,以及基因号,如下图所示:


下滑看到蛋白质序列
然后将蛋白质序列复制黏贴到记事本上(大家有什么更好的方法,评论区分享以下哈),格式如下:
>后面的注释是该序列的名称,可以根据需求更改
序列查找完成后先保存为txt格式,重命名更改为fasta格式;
2.建树前的准备-序列比对,点击 MEGA 主窗口上的 File → Open A File →找到并打开 *.fasta,将出现以下窗口:
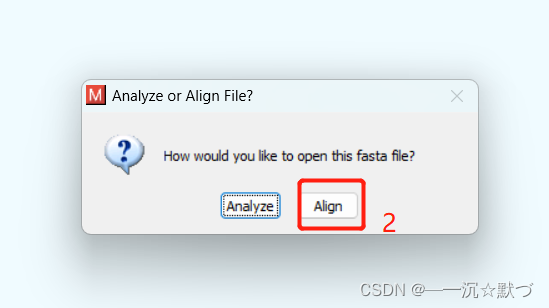

选择Align by ClustalW
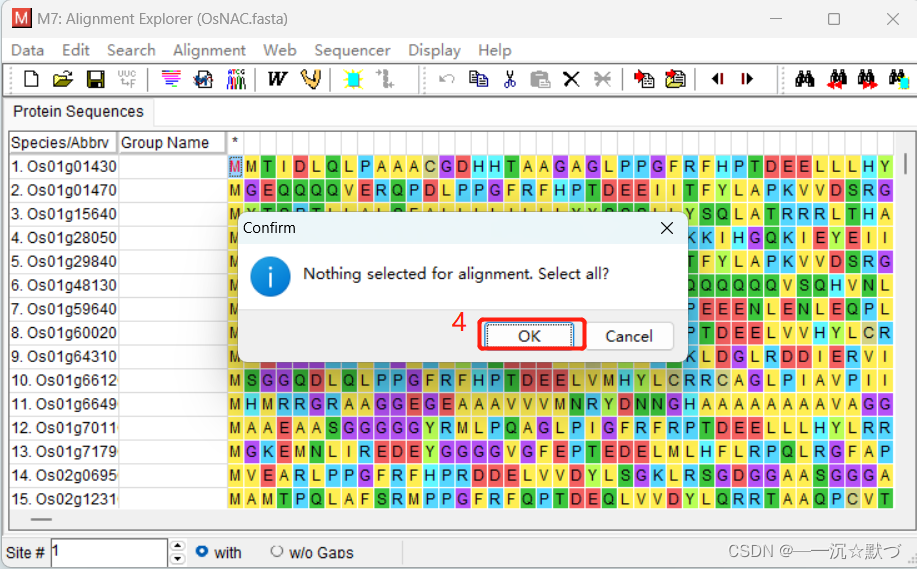
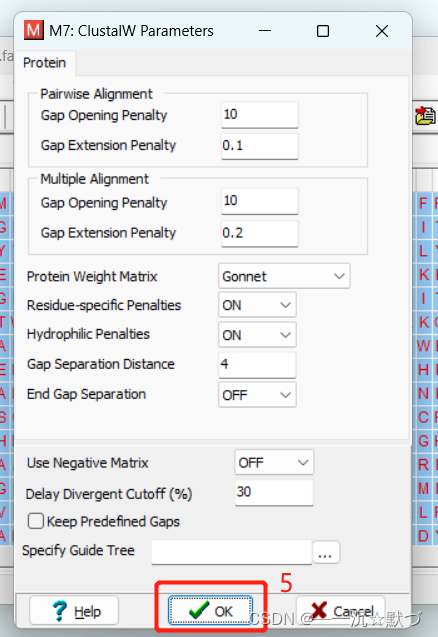
选择默认设置,点击OK生成比对文件
选择Data→Export alignment→MEGA Format
生成系统发育树,在这里选择的是最近邻居法,点击Phylogeny→Construct /Test neighbor-joining tree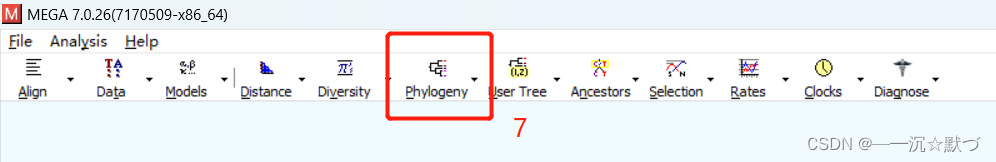
选择*.meg文件

生成系统发育树
根据需求更改形状

Ctrl+S保存,Export current tree(Newick)
方便之后导入iTOL进行美化
iTOL的使用下次再写哈,欢迎在评论区讨论
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!